Pour bien répondre aux questions, vous pouvez utiliser votre cours et les cours suivant, en cliquant sur le lien vous déclencherez le téléchargement d'un pdf :
- Cours du Professeur J. SAMARUT
- Cours du Professeur Joël LUNARDI
Proposition de plan de cours
Introduction et définitions
différents niveaux de régulation
définitions importantes :
Certaines séquences sur l' ADN sont responsables d'activation ou d'inhibition de la transcription des gènes par l'ARN polymérase ;elles sont appelées éléments cis- régulateurs ; cis signifie du même coté ou sur la même molécule.
Ici cela signifie que ce sont des portions d'ADN (séquences) qui participent à la régulation de la transcription.
Les partenaires obligatoires des séquences cis-régulatrices sont des protéines appelées facteurs de transcription ou éléments trans (trans = à coté ou en face) ; ces protéines activent ou inhibent la transcription des gènes, respectivement en stabilisant ou déstabilisant la fixation de l'ARN polymérase. Un facteur de transcription est une protéine qui est impliquée dans :
- l'initiation de la transcription en permettant la reconnaissance par l'ARN polymérase des séquences du promoteur (séquence Tata box par exemple) ;
- la régulation de la transcription, par exemple les facteurs trans-activateurs (qui se fixent sur les séquences enhancer ) et les facteurs trans-inhibiteurs (qui se fixent sur les silencers) ; N.B. chez les procaryotes on parlera plutôt de répresseurs et d'activateurs.
- la terminaison de la transcription.
séquence cis et facteurs trans ... en vidéo
les différents niveaux :
L'activation ou l'inhibition de la transciption d'un gène est le
résultat de l'interaction de différents facteurs à tous les niveaux
de complexité de l'organisme : de la molécule d'ADN, probablement
jusqu'au psychisme …
Les différents facteurs (pas tous connus) seraient :
- présence de séquences régulatrices à proximité ou à portée du promoteur (action à distance) ;
- modification chimique de l'ADN et des histones ; effet sur la compaction de la chromatine (épigénétique);
- maturation différentielles des ARNm eucaryotes ; épissage alternatif ; polyadénylation différentielle ;
- stabilité des ARNm dans le cytoplasme ;
- interférence à ARN ;
- stabilisation ou destabilisation des protéines ;
La régulation des gènes procaryotes
opérons et gènes solitaires chez les eubactéries
Les gènes simples à expression constitutive
gènes constitutifs
Un gène minimal procaryote possède au minimum un promoteur pour initier sa transcription, un RBS et une séquence codante (ORF = CDS : si c'est un gène de protéine) et une séquence de terminaison. Un gène à expression constitutive est transcrit en permanence (mais pas forcément à un taux élevé) ; sa transcription est en principe non régulée ; la transcription étant coûteuse en énergie, seuls les gènes codant des ARN de structure ( ARNt, ARNr chez tous les êtres vivants et les organites, snARN et ARNi chez les eucaryotes ...) et des protéines indispensables au métabolisme de base sont issues de la transcription de gènes constitutifs. En génie génétique, les gènes de resistance à un antibiotique sont constitutifs.Remarques :
- dans l'opéron lactose on inclut parfois le gène lacI qui code le répresseur de l'opéron lactose ; en fait, il ne fait pas partie de l'opéron lui même : lacI est un gène simple (ne contenant qu'une seule ORF , et il n'est donc pas régulé comme l'opéron lactose ;
-
les gènes AmpR (qui code une béta- lactamase qui hydrolyse l'ampicilline et rend donc la bactérie insensible à cet antibiotique), KanR
(contre la Kanamycine et d'autres aminosides), TetR (contre la tétracycline), etc. ; sont constitutifs et utilisés en génie génétique comme marqueurs de transformation et portés par la plupart des vecteurs plasmidiques utilisés.
Les opérons
le modèle lactose
opéron lactose
Historiquement c'est l'étude de ce groupe de gènes par Jacob et Monod (prix Nobel) qui a permis pour la première fois de comprendre comment les gènes bactériens sont régulés.
opéron lactose sur Wikipédia
les autres opérons
anabolisme, catabolisme et métabolisme secondaire
L'anabolisme correspond à l'ensemble des réactions biochimiques impliquées dans la synthèse ou fabrication des molécules biologiques. Le catabolisme correpond à l'ensemble des réactions biochimiques impliquées dans la simplification ou destruction des molécules biologiques (par exemple la digestion). Les métabolites secondaires sont produits de manière non essentielle par les êtres vivants lorsque les réactions du métabolisme de base fonctionnent correctement et que les cellules arrêtent de se diviser : ces substances sont souvent des poisons destinés à protéger les cellules des prédateurs ; chez les plantes ce sont principalement des tanins et des alcaloïdes ; chez les micro-organismes se sont par exemple les antibiotiques qui servent à contrôler les espèces concurantes.opérons impliqués dans le catabolisme
opérons du catabolisme
Le cas des opérons lactose, saccharose, xylose, ...
Tant que le glucose est présent en abondance dans le milieu, il est inutile pour la cellule et très coûteux en énergie de produire des enzymes inutiles dans ce contexte (béta-galactosidase par exemple). Quand le glucose est abondant le taux d'AMPc dans le cytoplasme bactérien est très bas. Quand le glucose devient rare l'adénylate cyclase est activée et elle transorme de plus en plus d'ATP en AMPc. Ce métabolite est appelé second messager ; c'est une substance qui est produite dans la cellule en cas de stress métabolique (manque d'énergie). L'AMPc peut se fixer sur différentes protéines effectrices (qui font des choses) comme la protéine CAP. La protéine CAP activée par la fixation de l'AMPc se fixe alors en amont de différents promoteurs liés au catabolisme comme les opérons permettant l'utilisation d'oses (sucres) plus rares et complexes que le glucose. Par exemple, en amont de l'opéron lactose existe un site de fixation du complexe CAP+AMPc qui augmente d'un facteur 50 la transcription de l'opéron lactose, mais celui-ci n'est vraiment totalement actif qu'en présence d'un inducteur (allo-lactose ou bien en génie génétique l' IPTG qui en se fixant sur le complexe du répresseur (tétramère dela protéine codée par le gène lacI) le déforme et l'empèche de se fixer sur l'opérateur de l'opéron lactose. En d'autres termes l'inducteur (IPTG) inhibe l'inhibiteur (répresseur) ; moins moins égal plus ; la transcription a lieu quand l'inducteur empèche le répresseur de bloquer l'opérateur.

opérons impliqués dans l'anabolisme
opérons de l'anabolisme
Le cas de l'opéron tryptophane
Site complet sur le sujet : cliquer ici
Produire en trop grande quantité un acide aminé rare et
complexe comme le tryptophane est trop coûteux en énergie,
l'anabolisme du tryptophane nécessite la synthèse de cinq protéines
différentes (5 enzymes). Á l'opposé du fonctionnement de
l'opéron lactose (qui est lié au catabolisme), c'est quand il y a
trop de tryptophane que l'opéron est réprimé.
Sources Wikipedia :
- en français
- en anglais (mieux !)
La régulation des gènes eucaryotes
gènes eucaryotes
Enhancer et silencer
L'expression des gènes eucaryotes est régulées de manière complexe à différentes échelles. Les séquences régulatrices se trouvent pour la plupart à proximité du promoteur et sont les points d'ancrage des facteurs de transcription qui activent (en se fixant sur les enhancer) ou inhibent (en se fixant sur les silencer)la transcription (donc la fixation de l'ARN polymérase sur le promoteur).
Structures des facteurs trans : facteurs de transcription
Toutes les molécules appelées facteurs de transcription sont des protéines. Elles possèdent toutes au moins deux domaines fonctionnels : c'est à dire que dans leur structure 3D, il est possible de distinguer deux parties ayant des rôles différents :
- le binding domain = BD ou domaine de
liaison à
l'ADN contient des acides aminés dont les chaînes latérales
établissent de nombreuses liaisons hydrogène (LH) avec
les bases
de la séquence d'ADN reconnue.
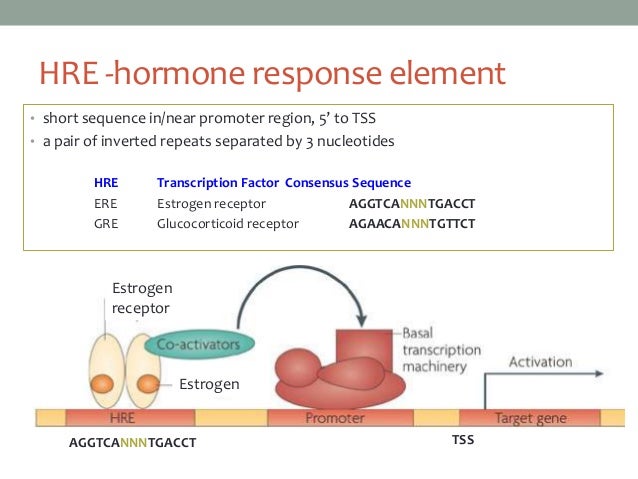
Plus la séquence reconnue est proche d'une séquence idéale appelée séquence consensus plus le nombre de LH est grand et plus la protéine (facteur trans) est solidement fixée sur la séquence cis (ou responsive element = RE).
Par exemple, le dimère Jun/Fos est constitué de deux protéines différentes qui s'associent pour former un facteur de transcription impliqué dans la régulation de la division cellulaire. Les gènes qui les codent sont appelés des oncogènes car lorsque jun et fos sont surexprimés à la suite d'une mutation (cancérigène), les cellules se divisent sans régulation. - le domaine trans-activateur (souvent riche en charge négatives qui stabilisent l'ARN polymérase plutôt basique ; c'est à dire positive) ou trans-inhibiteur (qui destabilisent l'ARN polymérase)
Image : séquences consensus de différents facteur de transcription

exemple de facteur de transcription homodimérique
exemple de la complexité de la région régulatrice

Technologies d'étude de l'expression des gènes
northern blot, puces à ADN, western blot, RT-PCR
Ce chapitre va traiter des méthodes permettant d'étudier qualitativement et éventuellement quantitativement l'expression des gènes. Ces techniques permettent de mesurer les effets de changements dans l'environnement des cellules sur leur phénotype
Mesure de la quantité d'ARNm produits
dos-blot, northern blot, puces à ADN, RT-PCR
RT-PCR
N.B. : sera vu en deuxième année. En résumé :
- les ARN totaux doivent être purifiés, l'ADN génomique détruit par hydrolyse enzymatique (DNase) pour ne pas interférer avec les mesures.
- les ARNm sont transcrits en sens inverse en ADNc à l'aide d'une amorce d'ADN et d'une transcriptase inverse (ADN polymérase ARN dépendante) appelée en anglais RT;
- l'ADNc est amplifié lors d'une PCR quantitative en temps réel qui permet de mesurer la quantité d'ADNc, donc d'en déduire la quantité d'ARNm produit dans l'échantillon analysé. Un étalonnage est donc nécessaire.
Techniques basées sur l'hybridation moléculaire : utilisation de sonde
Une sonde moléculaire est une molécule capable de détecter visuellement le partenaire avec lequel elle va se lier spécifiquement. Dans le cas des acides nucléiques, la sonde moléculaire sera un acide nucléique (ADN le plus souvent, ou ARN, ou PNA) dont la séquence est complémentaire inversée de la séquence à mettre en évidence et marquée de manière froide ou chaude.Marquage des sondes moléculaires en BMGG
Les sondes dites chaudes sont des sondes rendues radioactives par l'incorporation le plus souvent d'un phosphore 32 (isotope radioactif du phosphore noté 32P) dans sa molécule. La révélation de la sonde se fera par autoradiographie ou par mesure de scintillation ; cette méthode très sensible est souvent remplacée par une méthode froide moins problématique ...
Les sondes dites froides sont marquées :
- soit par un ligand détectable : antigène ou biotine ; une molécule reconnaîtra spécifiquement ce ligand et sera soit fluorescente, soit liée à une enzyme chromogénique.
- soit directement à un fluorochrome (cf. FISH painting), soit directement à une enzyme chromogénique ou luminogénique.
Mesure de la quantité de protéines produites
western blot, ELISA, immuno-précipitation et SDS-PAGE
Comparaison Western et Northern Blot
La technique inventée par le docteur Southern pour détecter une séquence spécifique au sein d'un mélange de fragments d'ADN fut adaptée respectivement à :
- la détection d'une séquence d'ARN au sein d'un mélange de molécules d'ARN de tailles différentes ; cette technique est appelée Northern Blot
- la détection d'une protéine au sein d'un mélange de protéines de tailles différentes ; cette technique est appelée Western Blot
points communs
Southern, Northern, et Southern Blot partagent trois étapes :
- la première consiste à séparer les molécules du mélange par électrophorèse ;
- la seconde consiste à transférer les molécules du gel vers une membrane qui fixe les molécules sans changer leur position relatives dans le gel ; ce trasfert se fait soit par capillarité soit par électrophorèse perpendiculaire ...
- la dernière consiste à révéler la présence des cibles recherchées après avoir saturé la membrane dans ses sites non spécifiques pour éviter des faux positifs ; cette dernière étape necéssite de nombreux lavages à chaque étape pour éliminer les molécules fixées non spécifiquement (évite les bruits de fond).
différences
------------